DISEÑO DE OLIGONUCLEOTIDOS INICIADORES (Primers)
Autor: Diego Mauricio Riaño Pachón
General (1)
La selección de oligonucleótidos iniciadores es muy importante
en la
reacción en cadena de la polimerasa
o PCR (por sus siglas en inglés), en la hibridizaciòn
con sondas y en la secuenciación de ADN. De este paso depende
el éxito en el laboratorio.
Principales Criterios
| Tamaño |
Tamaño ideal: 20-25 nucleótidos de longitud
generalmente: 18-30 nucleótidos de longitud |
| Base en el extremo 3' |
Debe ser una G o una C |
| Temperaturas de fusión (Tm) |
50-65 ºC |
| contenido GC |
40-60% |
| auto-complementariedad |
Debe ser evitada
Para minimizar la formación de estructuras secundarias y los
dimeros de primer |
| Similaridad |
Debe tener un 100% de apareamiento con el molde |
Cebadores para PCR o para Secuenciación
PCR:
Cuando el objetivo a amplificar es un locus, la posición de los
iniciadores debe ser relativa a los codones de inicio y terminación.
Es recomendable que el cebador "forward" se encuentre mas
o menos a 35 pb del inicio de la secuencia que codifica, de la misma forma
el cebador "reverse" debe localizarse 35 pb despues de la región
que codifica.
SECUENCIACIÓN
Cuando el objetivo es obtener la secuencia de ADN es recomendable que los
cebadores se localicen por lo menos 50 pb antes de la región que
se desea secuenciar, esto para el cebador "forward" cuando hay uno, y 50
pb despues de la región a secuencias para el cebador "reverse",
cuando hay uno.
Es importante que el fragmento a secuenciar no sea muy grande, debido
a que los errores de lectura de secuencia aumentan cuando los fragmentos
son grandes, si es necesario secuenciar un fragmento muy largo, es
conveniente
diseñar cebadores internos, para obtener la secuencia en dos partes
y luego ensamblarla.
Temperatura de Asociación
La temperatura de asociación de los cebadores es uno de los factores
mas determinantes de la reacción. Se recomienda que se emplee
como temperatura de asociación la temperatura de fusión -5ºC,
aproximadamente. Existen muchos métodos para calcular la temperatura
de fusión, pero siempre, despues de efectuado el cálculo
es necesario ir al laboratorio y ensayar con diferentes temperaturas de
asociación cercanas a la temperatura de fusión para determinar
la temperatura óptima para cada reacción.
Cálculo de la temperatura de fusión (Tm) (2)
Existen muchos métodos para estimar el valor de la temperatura de
fusión.
Para secuencias de 20 bp o menos, existe la siguiente aproximación:
Tm = 2ºC (A + T) + 4ºC (G + C)
donde se asume una concentración de sal de 0.9M, tipica de "dot
blot" y otros ensayos de hibridización
A continuación tenemos otra expresión para el calculo
de Tm
Tm = 81.5 + 0.41(%GC) - 500/L + 16.6 log[M],
donde L se refiere a la longitud del oligonucleótido, y [M] es
la concentración de cationes monovalentes. Esta
fórmulas es
unicamente aplicable a asecuencias polinucleotidicas largas.
El método mas preciso para estimar la Tm de oligonucleótidos
está basado en el análisis termodinámico del proceso
de fusión del cual se puede mostrar que,
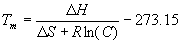
Los cambios en la entalpia(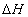 )
y la entropia(
)
y la entropia(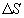 )
de la formación del duplex se calculan a partir de parametros termodinámicos
de vecindades. R es la constante molar de los gases (1.987 cal.K-1mol-1),
y C es la concentración molar del oligonucleotido. Se puede adicionar
un secundo término a la ecuación anterior para tener encuenta
el efecto estabilizante de la sal sobre el duplex:
)
de la formación del duplex se calculan a partir de parametros termodinámicos
de vecindades. R es la constante molar de los gases (1.987 cal.K-1mol-1),
y C es la concentración molar del oligonucleotido. Se puede adicionar
un secundo término a la ecuación anterior para tener encuenta
el efecto estabilizante de la sal sobre el duplex:
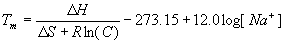
Los efectos de Na+ y K+ son equivalentes dentro de los margenes del
error experimental.
Programas de Diseño de Primers
Primer3
Primer Selection
NetPrimer
Bibliografía
1. -
Human Genome
Maping Project. Primer
Desing (http://www.hgmp.mrc.ac.uk/MANUAL/faq/faq-primers.html)
2. - Integrated DNA Technologies.
Calculation
of Tm for Oligonucleotides (http://www.idtdna.com/html/tech/tmcalc.html)


